近日,珠江水產(chǎn)研究所葉星研究員團隊完成了大口黑鱸染色體水平基因組精細圖譜的繪制和遺傳解析,研究論文“Chromosome-level genome assembly for the largemouth bassMicropterus salmoidesprovides insights into adaptation to fresh and brackish water”正式發(fā)表在國際重要學術(shù)期刊《Molecular Ecology Resource》上。
大口黑鱸(Micropterus salmoides)自然分布于北美淡水流域,但在海岸咸淡水環(huán)境中也有分布,是北美水生態(tài)系統(tǒng)中具有重要生態(tài)學與經(jīng)濟學意義的黑鱸屬重要成員之一。自1983年引進以來大口黑鱸已成為我國重要的養(yǎng)殖魚類,2019年我國養(yǎng)殖產(chǎn)量達47.8萬噸。為更好地了解大口黑鱸的物種進化史、開展其重要性狀遺傳基礎(chǔ)的研究以及種質(zhì)資源開發(fā)與利用,該團隊開展了大口黑鱸基因組研究。結(jié)合PacBio單分子實時測序與Hi-C技術(shù),獲得了首個高質(zhì)量染色體水平的大口黑鱸基因組。大口黑鱸基因組大小為964Mb,contig N50和scaffoldN50分別為1.23Mb和36.48Mb。結(jié)合PacbioRNA測序一共注釋出23,701個基因。
多數(shù)鱸形目魚類單倍型染色體數(shù)量為24條,而大口黑鱸卻僅有23條。本研究通過比較基因組分析發(fā)現(xiàn)其1號染色體為融合染色體的證據(jù):大口黑鱸的1號染色體分別對應(yīng)于花鱸的20和24號染色體、歐洲鱸的18-21和24-25號染色體,同時在大口黑鱸1號染色體的中上部24Mb處發(fā)現(xiàn)了高密度端粒元件區(qū)域,提示大口黑鱸1號染色體由2條端部著絲粒染色體融合所成,其中一個著絲粒可能丟失或退化。
論文還探討了大口黑鱸的咸淡水適應(yīng)的分子機制。通過收縮與擴張分析、正選擇分析和轉(zhuǎn)錄組分析,共發(fā)現(xiàn)大口黑鱸與離子調(diào)控相關(guān)基因294個。其中大口黑鱸閉合蛋白基因明顯擴張,產(chǎn)生27個家族成員共68個拷貝,該家族成員在調(diào)節(jié)細胞間緊密連接,離子與水的進出、維持滲透壓穩(wěn)定中發(fā)揮重要作用。大口黑鱸基因組精細圖譜的完成,為其種質(zhì)資源保護與改良、進化歷史及重要性狀的遺傳研究提供了重要的數(shù)據(jù)基礎(chǔ)。
該期刊為生物化學與分子生物學、生態(tài)學領(lǐng)域國際頂級期刊,中科院JCR分區(qū)生物學1區(qū)(Q1),IF為6.286。該研究受現(xiàn)代農(nóng)業(yè)產(chǎn)業(yè)技術(shù)體系專項資金(CARS-46)和水科院科技創(chuàng)新團隊項目的支持。中國水產(chǎn)科學研究院珠江水產(chǎn)研究所為論文第一完成單位,孫成飛博士與李佳博士為論文共同第一作者,葉星研究員和石瓊研究員為論文共同通訊作者。
文章鏈接:https://onlinelibrary.wiley.com/doi/epdf/10.1111/1755-0998.13256

Fig. 1The chromosome-level genome assembly and circos atlas of the largemouth bass.
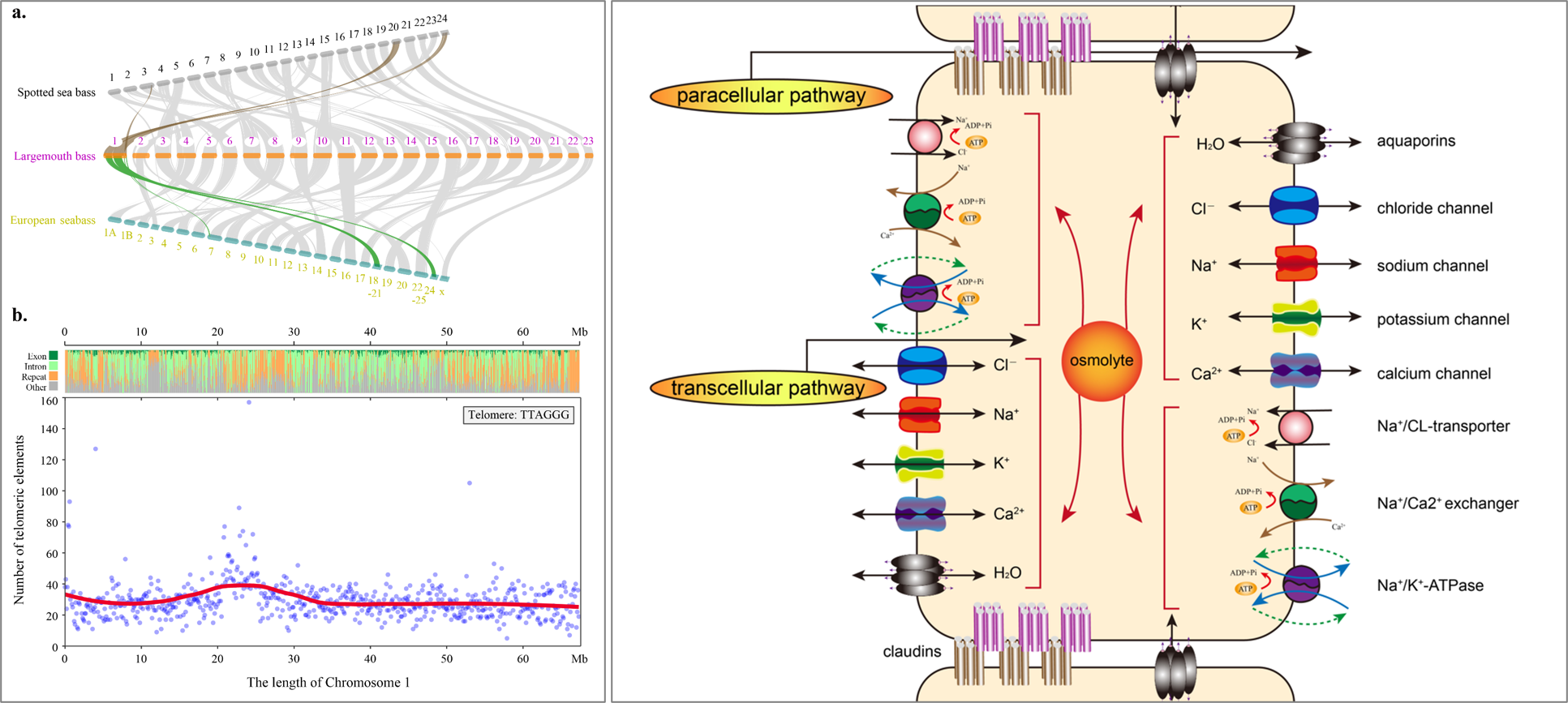
Fig. 2Chromosomal fusion in the Chr1(Left)and the proposed gene network map for ion regulation in LMB(Right)


